myPresto の最新版をダウンロードするページです。myPresto は、医薬品開発支援のために作成された分子シミュレーション計算のプログラム集です。
Documents for programs of myPresto5 in Japanese language.(Updated on September 26, 2018)
目次
簡易グラフィック分子設計ソフト
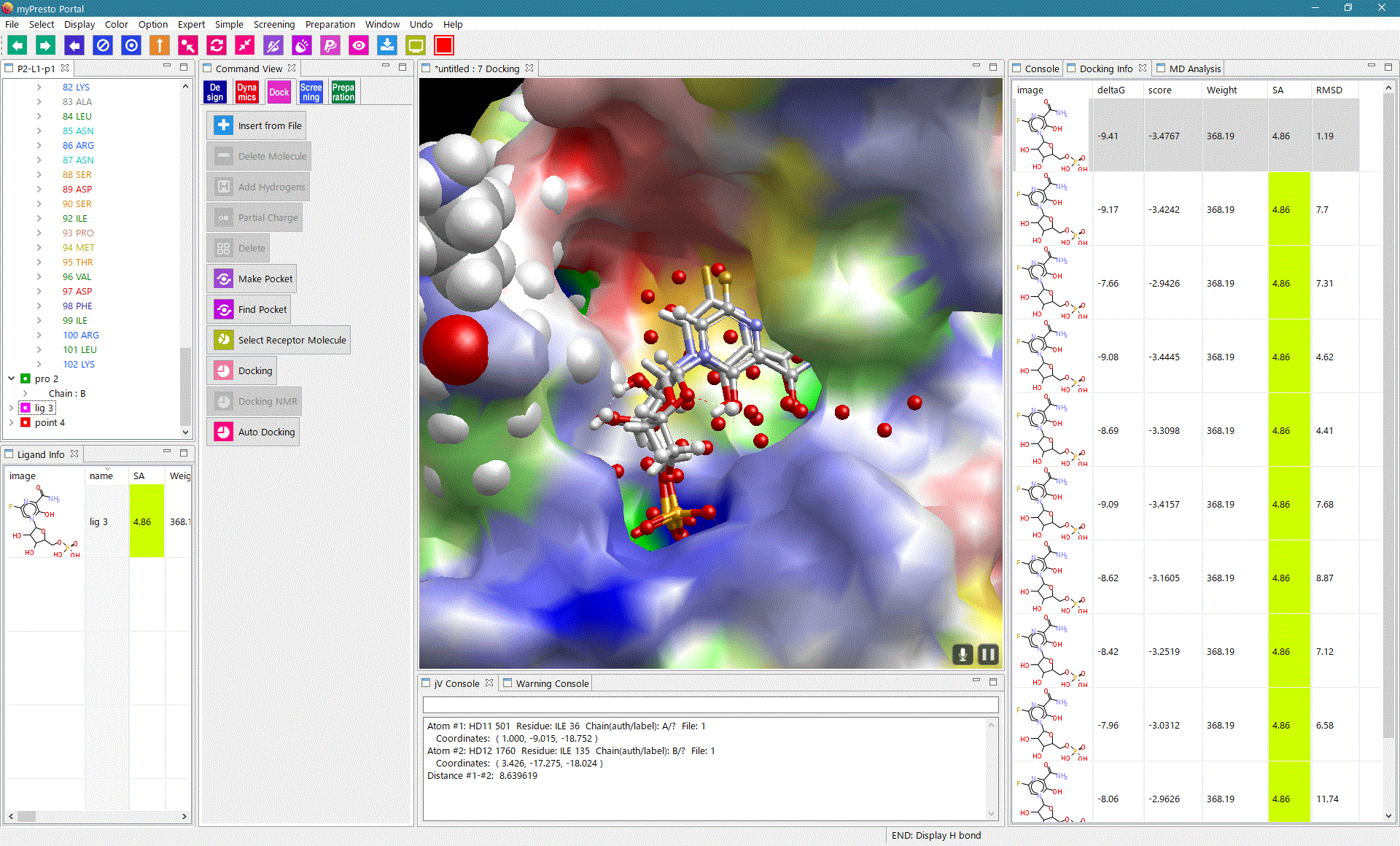
myPresto Portal
myPresto Protal は、myPresto と GROMACS のグラフィック・ユーザー・インターフェース (GUI) プログラムです。Windows, MAC, Linux (いずれも 64bit)で使用できます。 myPresto Portal tutorial (Japanese) (For ver. 1.1.60, updated on December 29,2018) |
Easy myPresto
Easy myPresto は、myPresto のグラフィック・ユーザー・インターフェース (GUI) プログラムです。Windows, MAC, Linux (いずれも 64bit)で使用できます。 |
| 無償版は、会員企業の厚意により有償ソフトに分子量や機能などの制限を加えて配給しています。 | |
| MolDesk EC ショップ | Easy myPresto 有償版 |
MDシミュレーション
cosgene: COnformation SamplinG ENginE (分子構造探索エンジン)
このエンジンは、マルチカノニカル分子動力学等、最新の機能を持つ分子動力学 (MD) ツールによって、膜蛋白質や低分子化合物との複合体を含む生体高分子系に対して、高能率の構造探索や自由エネルギー計算、結合自由エネルギーの算出を行うためのものです。Smooth-reaction path generation (SRPG) 法が結合自由エネルギーの計算手法として利用できます。
Fukunishi, Y., Mikami, Y., Nakamura, H. (2003) J. Phys. Chem. B. 107, 13201-13210
Last update on January 19, 2020
psygene-G: Protein dYnamics SimulatinG ENginE for GPU (分子動力学エンジン-GPU版)
このエンジンは、オリジナルな非エバルト法(Zero-Multipole summation法)を用いた静電相互作用計算による分子動力学 (MD)計算を、GPUを用いて高速に実施するためのものです。MPIによって複数のGPUによる並列化計算が容易に実行できます。
Mashimo, T., Fukunishi, Y., Kamiya, N., Takano, Y., Fukuda, I., Nakamura, H. (2013) J. Chem. Theory Comput. 9, 5599-5609
Last update on February 23, 2020
omegagene: a GPU-accelerated molecular dynamics simulator with enhanced conformational sampling
GPUで加速された分子動力学計算によって、より効率の良い構造探索が行える新たなエンジン omegagene がmyPrestoの一員に加わりました。非エバルト法(Zero-Multipole Summation method: ZMM) アルゴリズムを搭載し、改良したマルチカノニカル分子動力学 (v-McMD)法が利用できます。このプログラムは主に大阪大学にて開発され、オープンソース・プログラムとしても http://www.protein.osaka-u.ac.jp/rcsfp/pi/omegagene/ から利用いただけます。
Kasahara, K., Ma, B., Goto, K., Dasgupta, B., Higo, J., Fukuda, I., Mashimo, T., Akiyama, Y., Nakamura, H. (2016) Biophys. & Physicobiol. 13, 209-216: DOI 10.2142/biophysico.13.0_209
Last update on January 14, 2018
PDBcheck
入力PDBファイルの妥当性のチェック、タンパク質のN末・C末端の修飾、ジスルフィド結合(S-S)の判定、金属結合部位のアミノ酸のH化状態の編集などを行います。
setwater
タンパク質、DNA/RNA、膜、低分子を含む系に対し、溶媒水分子を球状(孤立系)、直方体形(周期系)で付加します。
add_ion
タンパク質、DNA/RNA、膜、低分子を含むsetwaterで水和した孤立・周期系に対し、指定した濃度でNaClによる電荷の中和、任意の塩濃度に調整します。
GROMACS対応ツール
convertTpl、ctl2mdp.pl、log2form.pl
タンパク質、核酸、低分子を含む系の作成はmyPrestoでは簡単です。myPrestoで作成した系のファイルを高速MDソフトGromacsの入力に
変換、またGromacsの出力ファイルを可読性の良いmyPresto形式のファイルに変換します。
低分子等の構造準備・ファイル書式変換
Hgene: Hydrogen Generation ENginE (水素原子構築エンジン)
有機低・中分子に対して、pH7付近での解離形・非解離形での水素原子の付加・削除、簡易な3次元構造発生、Gasteiger電荷、MOPAC AM1BCC電荷の付与、MOPAC PM3での構造最適化などができます。
対応書式は、SMILES / ISIS MOL, SDF / Sybyl mol2 / PDB / mmCIF
Last update on March 23 2022
力場パラメータ割り付けソフト
tplgeneX: ToPoLogy generatinG ENginE eXtended(高分子トポロジー構築エンジン)
このエンジンは、myPrestoの他のプログラムで利用する生体高分子の分子トポロジーを構築するためのものです。新たにPDBx/mmCIFフォーマットによる原子座標の入出力を可能にしたほか、リガンド分子のトポロジーの扱いも簡略にしました。
対応分子:タンパク質(リン酸化・メチル化含む)、核酸(RNA / DNA)、膜分子、水、各種塩、代表的溶媒(DMSO, 単糖類等)、金属、など
対応力場:
AMBER力場:ff99, ff99-Fuji, ff99-SB, ff99-SB-ILDN, ff99-SB-DISP
核酸力場:ff99, ff99bsc0, OL3, YIL, ROC, Shaw
膜分子力場: lipid14
Last update on March 23, 2022
tplgeneL: ToPoLogy generatinG ENginE for Ligands (リガンド分子トポロジー構築エンジン)
このエンジンは、myPrestoの他のプログラムで利用するリガンドの分子トポロジーを構築するためのものです。
Last update on January 12, 2018
膜系作成ツール
membgene3
membgene3は、膜貫通タンパク質・脂質二重膜・水・イオンを含む系の座標を作成するためのツールプログラムです。
Last updated on October 2 2021
自由エネルギー計算
Filling potential method
Filling potential法は、反発ポテンシャルと求心ポテンシャルを与えたUmbrella Potentialを使ったMD計算を多数実行して、使用したUmbrella Potentialを考慮して、座標トラジェクトリ群のヒストグラム解析を行うことによって、2つの分子の結合自由エネルギーを計算します。
Last updated on March 11 2018
配座発生ツール
Confgene:Conformer Generation ENginE(コンフォーマ構築エンジン)
( ToolsYYMMDD.tar.gzに梱包されています。)
低分子リガンドのコンフォーマ構造を発生します。
Last update on January 18, 2018
ConfgeneC: Conformer Generation ENginE for Cyclic part
(環状分子のコンフォーマ構築エンジン, ToolsYYMMDD.tar.gzに梱包されています。)
低分子リガンドにおける環状部分について、コンフォーマ構造を発生します。
Last update on January 18, 2018
descgene
低分子の環配座・直鎖を含む配座をランダムに発生させ、分子記述子ファイルを生成します。
(慣性半径、ASA、扁平率、離心率、MACCS key/Joback記述子等)
化合物データベースと生成ツール
LigandBox: LIGANd Data Base Open and eXtensible (リガンド分子データベース)
LigandBoxは、ドッキングにすぐ使えるように、水素原子付加と部分電荷情報付加の処理をして、立体構造情報を持つ化合物データを含むデータベースです。LigandBoxには、web版LigandBoxと、スクリーニング用LigandBoxがあります。web版LigandBoxには、市販されている低分子化合物に加え、KEGG DRUG、KEGG COMPOUND、PDBのデータベースで提供されている低分子化合物が含まれています。スクリーニング用LigandBoxは、screening_packを使った薬剤候補化合物スクリーニングで使用するためのリガンド分子データベースです。MTS, ML-MTS, DSI法によるスクリーニング計算を実行する際に必要な相互作用行列データと一緒に提供されています。スクリーニング用LigandBoxには、市販されている低分子化合物の中から、フィルターリングされた化合物が含まれています。
・web版LigandBox
こちらのリンクからアクセスできます。
・スクリーニング用LigandBox
※ スクリーニング用LigandBox をご要望の方は、ご所属を明記の上、お問い合わせでお申し込みください。ダウンロードサイトをメールでご連絡いたします。
LigandBoxの分子ファイルの例
3DdataConstruction: (リガンド分子構造構築ツール)
低分子化合物の2次元電子カタログ(2D mol2ファイル)から3次元分子構造の3D mol2ファイルを構築するツールです。
Last update on December 3, 2019
蛋白質リガンド結合サイト探索
MolSite: (リガンド結合ポケット探索ツール)
低分子リガンド・ライブラリーを用いて、それらのリガンドをタンパク質にドッキングさせることによって、リガンド結合ポケットの探索を行います。
Y. Fukunishi, H. Nakamura. “Prediction of ligand-binding sites of proteins by molecular docking calculation for a random ligand library.” Protein Science, in press
Last update on February 26, 2019
sitegene
蛋白質・核酸の分子表面の凹みのある部分を探索します。
UAP: Univeral Active Probes
ドラッグライクな低分子リガンド・ライブラリです。UAPを用いることにより、active compoundsが未知の場合においても、ensemble dockingのためのタンパク質受容体の良いモデル構造を選択できます。
Y. Fukunishi, K. Ono, M. Orita, H. Nakamura. “Selection of in-silico drug screening result by using universal active probes (UAPs).” Journal of Chemical Information and Modeling, 2010, 50, 1233-1240
Last update on January 14, 2018
蛋白質ー化合物ドッキング
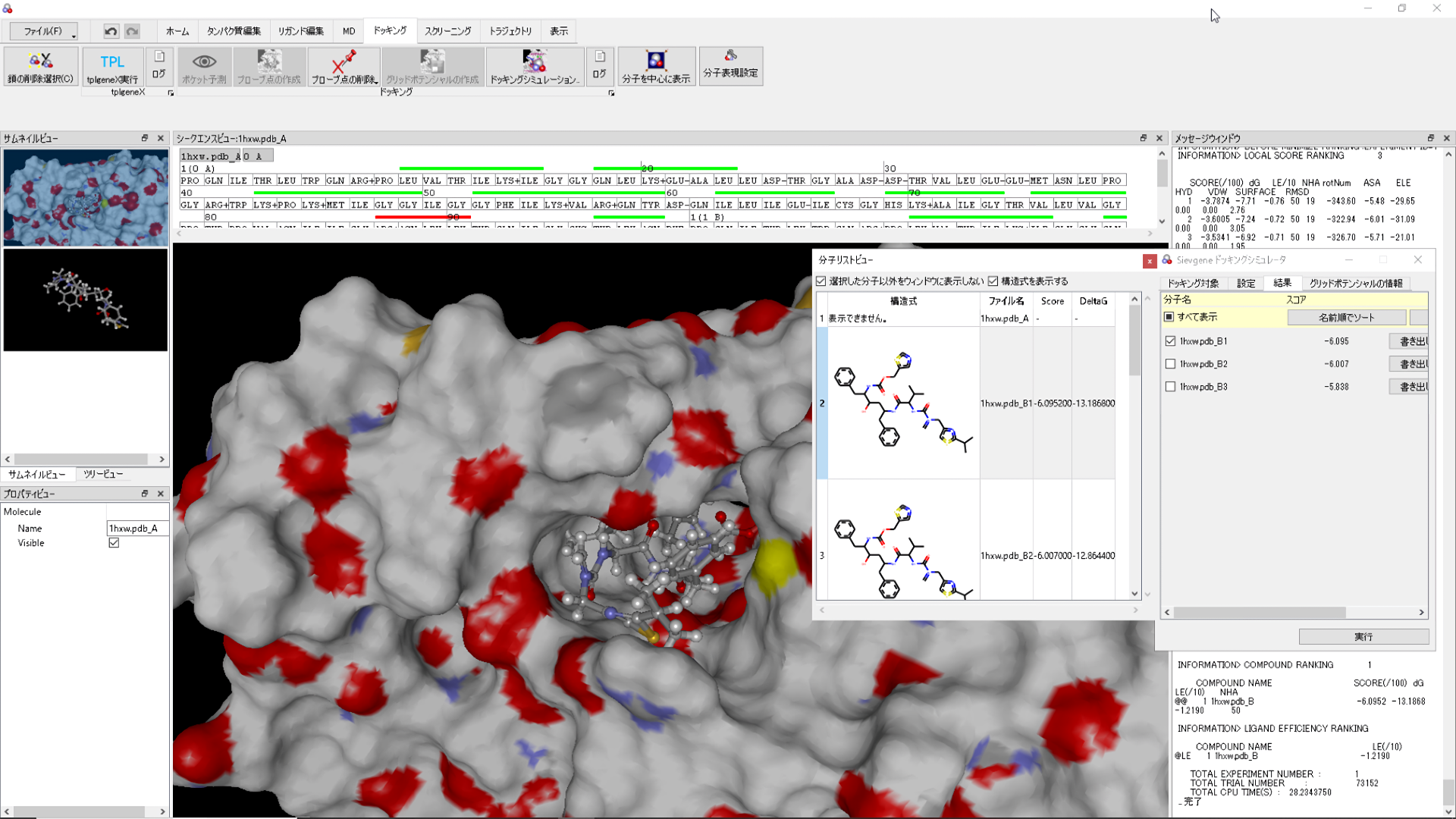
sievgene: SIEVinG ENginE (ドッキング・エンジン)
このエンジンは、フレキシブル・ドッキングの手法により、化合物を生体高分子へドッキングさせるためのものです。
Fukunishi, Y., Mikami, Y., Nakamura, H. (2005) J. Mol. Graph. Model. 24, 34-45
Last update on March 20, 2018
sievgene_M(ドッキング・エンジン)
sievgene_Mは、sievgeneの改良版です。sievgene_Mは、新しく登場したIntel社のメニーコアCPU(Xeon Phi)でも実行可能です。Intel Xeon Phiは、50個以上のコアを搭載したCPUです。
Last update on January 20, 2018
sievgeneDIAV: SIEVinG ENginE with DIAV (ドッキング+結合自由エネルギー推定)
このプログラムでは、上記sievgeneによるフレキシブル・ドッキングに加えて、蛋白質と化合物との結合自由エネルギーの値をさらに精度良く推定する(1.3 kcal/mol程度の誤差)ためのものです。
Fukunishi, Y., Nakamura, H. (2013) Pharmaceuticals 6, 604-622
Last update on May 7, 2022
sievgeneNMR: SIEVinG ENginE for NMR (ドッキング・エンジン NMR)
このエンジンは、NMRによる蛋白質―化合物相互作用のシグナル情報を取り込んだフレキシブル・ドッキングの手法であり、化合物を生体高分子へドッキングさせるためのものです。NMRシグナルの取り込みにより複合体予測精度は、約2倍に向上しています。
Fukunishi, Y., Mizukoshi, Y., Takeuchi, K., Shimada, I., Takahashi, H., Nakamura, H. (2012) J. Mol. Graph. Model. 31, 20-27
Last update on August 24, 2019
sievgeneMVO: SIEVinG ENginE for Maximum Volume Overlap (ドッキング・エンジン MVO)
このエンジンは、既存の蛋白質-化合物複合体立体構造のリガンド構造情報を取り込んだフレキシブル・ドッキングの手法であり、化合物を生体高分子へドッキングさせる、ないしスクリーニングするためのものです。ユーザーが与えた薬物ドッキングポーズへのテスト化合物の分子体積重ね合わせをドッキングと同時に行うことで、ユーザーのイメージした分子に近い化合物を選択できます。
Fukunishi, Y., Nakamura, H. (2012) Pharmaceuticals 5, 1332-1345
Last update on January 11, 2018
docking_score_QSAR
Docking-score QSAR法とは、与えられた低分子化合物と標的分子(蛋白質、核酸)との相互作用を既知活性情報を基にして回帰モデルにより予測する手法です。
Fukunishi, Y., Yamashita, Y., Mashimo, T., Nakamura, H. (2018) Molecular Informatics 37, 1868-1743
Last update on February 16, 2020
薬物スクリーニング
selectMTS: Multiple Target Screening method
(screening_packYYMMDD.tar.gz, cosgene_packYYMMDD.tar.gz等 に梱包されています。)
オリジナルのMTS法、 machine-learning MTS (MSM-MTS) 法、 direct score modification MTS (DSM-MTS)法の3つの手法が利用できます。もし、ターゲットとする受容体に対するactive compoundsが既知の場合には、 machine-learning MTS が最もヒット率が高い方法です。
Y. Fukunishi, Y. Mikami, S. Kubota, H. Nakamura, “Multiple target screening method for robust and accurate in silico screening.” Journal of Molecular Graphics and Modelling,25, 61-70 (2005)
Y. Fukunishi, S. Kubota, H. Nakamura, “Noise reduction method for molecular interaction energy: application to in silico drug screening and in silico target protein screening”, Journal of Chemical Information and Modeling, 46, 2071-2084 (2006)
Last update on October 29, 2018
selectDSI: Docking Score Index method
(screening_packYYMMDD.tar.gz に梱包されています。)
ligand-based drug screening法として、 オリジナルのdocking score index (DSI)法と、 machine-learning DSI (ML-DSI) 法が利用できます。もし、ターゲットとする受容体に対するactive compoundsが複数既知の場合には、 machine-learning DSI が高いヒット率を示せます。
Y. Fukunishi, Y. Mikami, K. Takedomi, M. Yamanouchi, H. Shima, H. Nakamura. “Classification of chemical compounds by protein-compound docking for use in designing a focused library”, Journal of Medicinal Chemistry, 49, 523-533 (2006)
Y. Fukunishi, S. Hojo, H.Nakamura, “An efficient in silico screening method based on the protein-compound affinity matrix and its application to the design of a focused library for cytochrome P450 (CYP) ligands.”, Journal of chemical information and modeling, 46, 2610-22 (2006)
Last update on January 12, 2018
Affinity_fingerprint: Compound database and protein-compound affinity matrix (タンパク質‐リガンド分子ドッキングスコア・マトリクス)
低分子リガンドと、182タンパク質に対する200万の低分子化合物のSievgeneによるドッキングスコア・マトリクスを提供します。このデータベースがselectMTSおよび selectDSIに使われます。このデータベースの入手先情報は、screening_packマニュアルに記載されています。
Substrucure_search
化合物の構造類似性に基づいてligand-based drug screeningを行なうプログラムです。Ullmanの方法により、低分子リガンドの立体構造に基づいて、類似したサブ構造探索を行います。
Last update on January 12, 2018
TGS: Topological Graph Search (A Similarity Search Using Molecular Topological Graphs)
Molecular Edge マトリクスの固有値に基づき、低分子リガンドの分子の類似性を解析します。
Y. Fukunishi, H. Nakamura. “A similarity search using molecular topological graphs”, Journal of Biomedicine and Biotechnology, Volume 2009 (2009), Article ID 231780
Last update on January 13, 2018
MVO: Molecular-Dynamics Maximum-Volume Overlap
分子動力学計算により、低分子リガンド同士の体積の重なりが最も大きくなるような配置を考えることによる、リガンド分子の類似性解析を行います。
( ToolsYYMMDD.tar.gzに梱包されています。)
Y. Fukunishi, H. Nakamura, “A new method for in-silico drug screening and similarity search using molecular-dynamics maximum-volume overlap (MD-MVO) method”, Journal of Molecular Graphics and Modelling, 2009, 27, 628-636
Last update on January 14, 2018
MVO_screening
MVO_scoreeningは、MVO(Maximum volume overlap)法を使って、クエリー分子と重なりが大きい分子を探索するプログラムです。
Last updated on February 27, 2018
分子物性推算
PropPred: (リガンドの物性値予測ツール)
回帰学習を用いた低分子化合物および中分子の物性値予測ツールです。学習済みデータを用いて膜透過係数(LogPa, apparent permeability)、電離を考慮した分配係数(LogD, distribution coefficient)、溶解度(LogS)の予測値を計算します。
Y. Fukunishi, T. Mashimo, T. Kurosawa, Y. Wakabayashi, H.K. Nakamura, K. Takeuchi. “Prediction of Passive Membrane Permeability by Semi‐Empirical Method Considering Viscous and Inertial Resistances and Different Rates of Conformational Change and Diffusion.” Molecular informatics, 2019:38, 1900071
Last update on January 30, 2022
LogS_prediction: (溶解度推定ツール)
分子記述詞と溶媒和自由エネルギーに基づいて低分子化合物の溶解度(LogS)を推定し、アグリゲータも予測します。
T. Mashimo, Y. Fukunishi, M. Orita, N. Katayama, S. Fujita, H. Nakamura. “Quantitative analysis of aggregation-solubility relationship by in-silico solubility prediction.” International Journal of High Throughput Screening, 2010:1, 99-107
Last update on January 14, 2018
合成容易性予測・分子エディター
ReCGen: Generate virtual compounds based on input structure
フラグメントベースのバーチャル化合物生成プログラムのソースコードです。
このプログラムはフラグメントDB作成ツール、DBマージツール、構造生成ツールの3つの機能を提供しております。
ReCGenは京都コンステラ・テクノロジーズが著作権を有する化合物構造生成プログラムです。
Last updated on March 1, 2018
SyntheticAccessibility: Prediction of the synthetic accessibility of given compounds
与えられた化合物の合成容易性を、分子の複雑度、光学活性中心、対称性から予測するプログラムです。合成容易性は、0(簡単)~10(困難)の数字で表示されます。
Y. Fukunishi, T. Kurosawa, Y. Mikami, H. Nakamura. Prediction of Synthetic Accessibility Based on Commercially Available Compound Databases. Journal of Chemical Information and Modeling. (2014), 54 (12), 3259-3267.
Last updated on January 11, 2018
PDBx/mmCIF書式対応ツール
LibMyPresto: Library package for parsing and writing of PDBx/mmCIF and PDB formatted files
PDBが提供するPDBx/mmCIFフォーマットとPDBフォーマットの双方のデータの読み書きを行うライブラリを提供します。C言語、f77およびf90のプログラムとリンクさせ、それらから呼び出して使う事ができます。